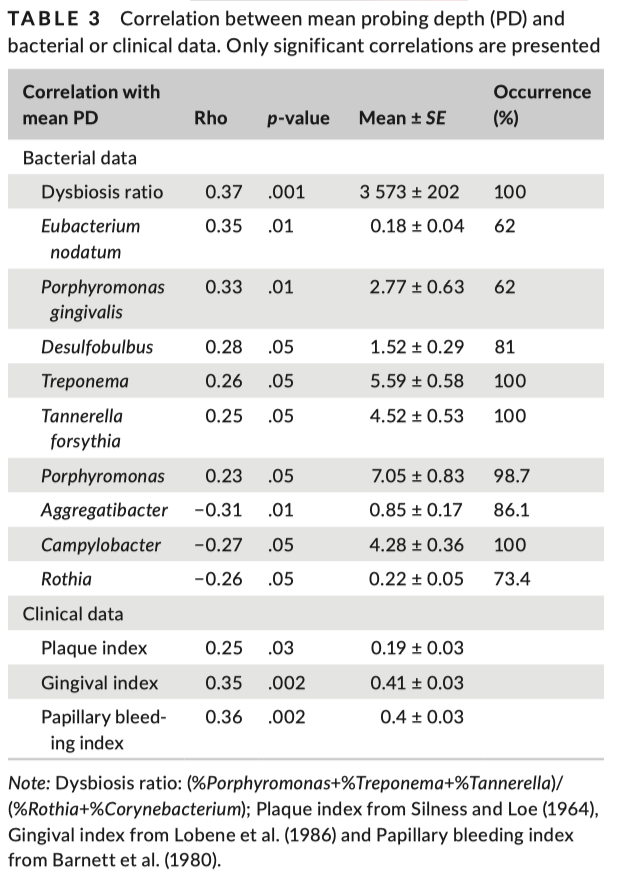
L’utilisation du séquençage et de la bioinformatique de nouvelle génération a révélé la complexité et la richesse du microbiote oral humain. Alors que certaines espèces sont bien connues pour leur pathogénicité parodontale, les approches moléculaires pour l’identification bactérienne ont sensibilisé à de nouveaux pathogènes parodontaux putatifs. Bien qu’ils se trouvent augmentés en cas de parodontite, il y a actuellement un manque de données sur leur interrelation avec les atteintes parodontales. Ici, les données de séquençage du microbiote sous-gingival issues de 75 patients atteints d’hémochromatose et de parodontite chronique ont permis de caractériser les pathogènes parodontaux sous-gingivaux. Des tests de corrélation et des modèles statistiques ont été mis en oeuvre pour évaluer l’association entre les pathogènes parodontaux et la profondeur moyenne des poches, et pour déterminer les biomarqueurs bactériens les plus pertinents de la gravité de la parodontite. Sur la base des résultats des tests de corrélation, neuf taxons ont été sélectionnés et inclus dans les modèles statistiques. Les modèles de régression linéaire multiple ajustés pour les variables cliniques systémiques et parodontales ont montré que la profondeur moyenne des poches était associée négativement à Aggregatibacter et Rothia, et positivement associée à Porphyromonas. De plus, un rapport bactérien précédemment décrit comme une signature de la dysbiose dans la parodontite (% Porphyromonas +% Treponema +% Tannerella) / (% Rothia +% Corynebacterium) était le prédicteur le plus significatif. Dans cette population spécifique, nous avons constaté que le meilleur modèle pour prédire la profondeur moyenne des poches était la dysbiose microbienne en utilisant la formule des taxons du ratio de dysbiose. Si des études complémentaires sernt nécessaires pour évaluer la validité de ces résultats sur la population générale, un tel rapport de dysbiose pourrait être utilisé à l’avenir pour surveiller le microbiote sous-gingival.
Emile Boyer, Bénédicte Martin, Sandrine Le Gall-David, Shao B. Fong, Yves Deugnier, Martine Bonnaure-Mallet, Vincent Meuric1
Periodontal pathogens and clinical parameters in chronic periodontitis
Mol Oral Microbiol. 2020;35:19-28. PubMed.
The use of next generation sequencing and bioinformatics has revealed the complexity and richness of the human oral microbiota. While some species are well known for their periodontal pathogenicity, the molecular-based approaches for bacterial identification have raised awareness about new putative periodontal pathogens. Although they are found increased in case of periodontitis, there is currently a lack of data on their interrelationship with the periodontal measures. We processed the sequencing data of the subgingival microbiota of 75 patients with hemochromatosis and chronic periodontitis in order to characterize the well-described and newly identified subgingival periodontal pathogens. We used correlation tests and statistical models to assess the association between the periodontal pathogens and mean pocket depth, and to determine the most relevant bacterial biomarkers of periodontitis severity. Based on correlation test results, nine taxa were selected and included in the statistical models. The multiple linear regression models adjusted for systemic and periodontal clinical variables showed that mean pocket depth was negatively associated with Aggregatibacter and Rothia, and positively associated with Porphyromonas. Furthermore, a bacterial ratio that was previously described as a signature of dysbiosis in periodontitis (%Porphyromonas+%Treponema+%Tannerella)/(%Rothia+%Corynebacterium) was the most significant predictor. In this specific population, we found that the best model in predicting the mean pocket depth was microbial dysbiosis using the dysbiosis ratio taxa formula. While further studies are needed to assess the validity of these results on the general population, such a dysbiosis ratio could be used in the future to monitor the subgingival microbiota.